Arbeitsgruppen (HU):
|
|
Spektroskopie, Elektrophysiologie, Kristallisation von Photorezeptoren, Optogenetik Vergabe von Bachelor- und Masterarbeiten
|
|
|
Virus Entry and Assembly, Cellular trafficking, Biomimetic nanoparticles, Membrane structure and dynamics, high resolution microscopy, Force spectroscopy/microscopy Vergabe von Bachelor- und Masterarbeiten
Nanotechnologie
|
|
|
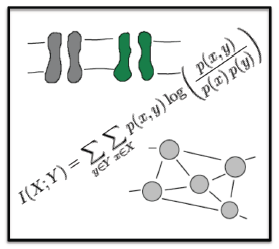
Mathematische Modellierung von zellulären regulationsnetzwerken wie Metabolismus, Signalweiterleitung oder Genregulation, theorie dynamischer Systeme, Fluoreszenzmikroskopie Vergabe von Bachelor- und Masterarbeiten
|
|
|
|
Assoziierte Arbeitsgruppen:
|
|
Charité Stat. und zeitaufgelöste FTIR und UV/Vis Spektroskopie, Photorezeptoren, Phytochrom, Kanalrhodopsine, GPCR´s Rhodopsin
|
|
|
HU, RKI Modellierung
|
|
|
MDC Modellierung
|
|
|
HU, Charité Theoretische Chronobiologie, Modelle der Genregulation
|
|
|
FMP Molekulare Pharmakologie und Zellbiologie
|
|
|
HU Neurobiologie
|
|
|
MPI IB Infektion, Kristallisation, Biophysik
|
|
|
HU Neurobiophysik
|
|
|
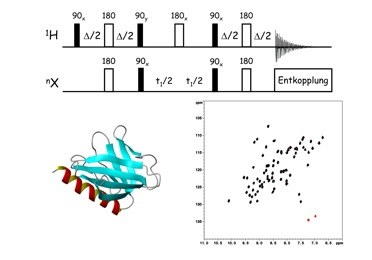
FMP NMR, Biocore, Fluoreszenz, ITC
|
|
|
FMP Electrophysiology, structural biology, unnatural amino acids, fluorescence
|
|
|
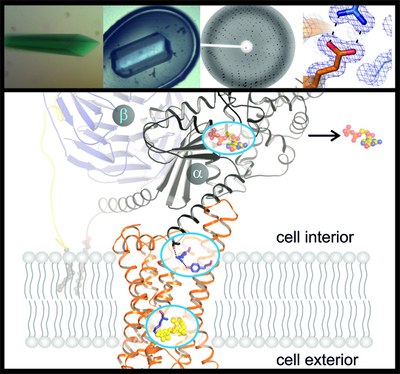
Charité Inhaltliche Schwerpunkte: Signaltransduktion, Membranproteine (wie z.B. G-Protein-gekoppelte Rezeptoren (GPCRs)), licht-sensitive Proteine (z.B. Photolyasen) und Photorezeptoren (z.B. Rhodopsine, Phytochrome), Metalloenzyme (z.B. [NiFe]-hydrogenase), Methoden: Heterologe Expression, Protein-Engineering, Protein-Reinigung und -Charakterisierung, Kristallisation und Röngtenstrukturanalyse von Proteinen, Spektroskopsiche Methoden an Proteinkristallen
|
|
|
FMP NMR
|
|
|
HU Neurophysiologie
|
|
|
Charité Kryo-Elektronenmikroskopie, Makromolekulare Komplexe und Maschinen, Proteinbiosynthese, Ribosomen
|
|
|
MDC Mathematische Modellierung von Signaltransduktionsprozessen, insbesondere des NF-kappaB und Wnt/ beta Catenin Pathways, sowie Genregulation Netzwerkanalyse unter normalen und krankheitsrelevanten Bedingungen
|
|
|